
NEWS
新闻中心
SERVERS
服务项目
发布时间:2021-03-26
核心摘要
再生是一种适应性现象,具有广泛的生物学意义,在包括人类在内的几乎所有有机体中广泛传播,不同物种的再生能力因其障碍而异。
斑马鱼尾鳍组织的表观形态因其具有离散和快速的再生能力而被广泛研究,一些基因/蛋白质被发现与斑马鱼尾鳍组织再生有关。在此,我们用一种新的CRSISPR-Cas9基因敲除方法评价了Annexin2a和2b基因在成年斑马鱼尾鳍组织再生中的功能作用。这两个基因单独被敲除会导致再生速度减慢,下调靶基因及其搭档基因/蛋白(如ANXA1a、ANXA5b和ANXA13)。本研究验证了一种新的基因靶向方法和Annexin在再生机制中的可能作用。

一、综述
真核膜联蛋白家族,由在脊椎动物中鉴定的11种蛋白质组成(ANXA1-7、9、11、13和31)。在斑马鱼中鉴定了4个基因(ANX1a、ANX2a、ANX5和ANX11a。全部ANXAs显示一个高度保守的内含子-外显子组织,并具有一个特征——高度保守的70-氨基酸结构域,重复4至8次,具有钙依赖性阴离子磷脂结合的能力(ANX31除外,ANX31缺乏钙结合位点)。然而,这种进化上保守的蛋白质家族的功能仍然知之甚少,ANXA5目前正在广泛研究斑马鱼活凋亡显像。通过许多研究,对该基因家族的进化史进行了研究,斑马鱼再生组织中ANX2a和2b表达较高。
1970年标志着生物学新时代的开始,重组DNA技术赋予了分子科学家操纵DNA的能力,直到今天,在细菌和古细菌中发现的一种适应性免疫系统都是简单的基因操纵平台之一。CRISPR-Cas将其先前遇到的病毒和质粒中的DNA片段标记为一系列CRISPR重复序列,并在以后用作引导CRIPSR RNA(crRNA)靶向该家族的新感染。
化脓性链球菌II型CRISPR系统被各种细菌用作灭活外源DNA的机制,一些报告也证实了这一点上斑马鱼与这种技术相一致。它的易用性、低成本,以及多路复用的可能性,使得它成为斑马鱼大规模反向遗传屏幕的主要工具。在许多情况下,基因功能在体内的研究需要基因的时空控制沉默。在这里,我们提出了斑马鱼组织特异性基因失活的原理,使用CRISPR/Cas9技术和详细的方法来实现它。
在我们早期工作的基础上,我们用转录组学鉴定了负责斑马鱼再生的Annexin基因。 我们决定对Annexin基因家族进行进一步的研究,因为斑马鱼和人Annexins之间有70%以上的同源性。为了研究这些靶基因的作用,我们设计了目前的研究,使用CRISPR来敲除ANXA2a和2b基因,并研究其在表型和基因型水平上的作用。
二、实验方法
1、用SGRNA构建表达载体:
利用ZiFiT靶向程序设计了ANXA2a和ANXA2b基因特异性的CRISPRsgRNA靶点。所有靶点均合成为寡核苷酸和反义寡核苷酸(Bioserve,印度)(图1a)。
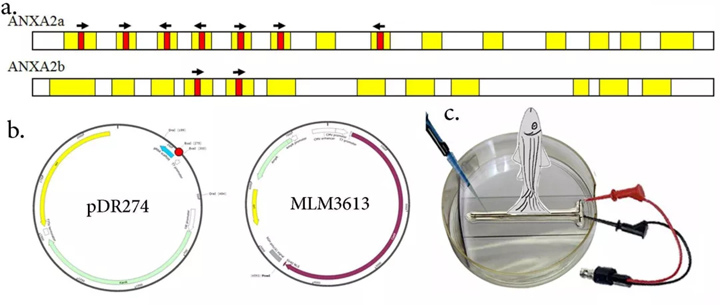
图1:1a.具有外显子(黄色阴影)和CRISPR-Cas目标位点(红色阴影)的ANXA2a和2b基因示意图,箭头指示CRISPRCas目标的方向。 b.DR274质粒图谱,红色点表示克隆位点和MLM3613载体。c.卡通详细介绍了CRISPR-Cas9RNA在成年斑马鱼截肢鳍组织中的电穿孔方案。
寡核苷酸对通过在95°C下孵育25个正向和反向寡核苷酸的pmols相互退火3min,然后将混合物的冷却降至30°C。从Addgene(质粒#42250)获得pDR274载体),用作合成sgRNA的表达载体ANXA2a和ANXA2b目标。用BSAI将100ngpDR274 载体线性化,用RAPID连接酶(Invitrogen)与退火寡核苷酸连接,用热休克转化协议在DH5α细胞中转化,并在LB卡那霉素选择板中选择阳性克隆(图1b)。用M13F和基因特异性引物对阳性克隆进行菌落PCR和测序。
2、SGRNA mRNA合成:
在DraI限制性消化后,合成了特异性于ANXA2a和ANXA2b靶的sgRNAmRNA,用于洗脱含有T7 启动子和sgRNA靶的282bp片段。根据制造商的协议,用MAXI script ® T7试剂盒(ThermoFischerScientific)对sgRNA进行了体外转录。然后用酸性苯酚氯仿和酒精沉淀纯化DNase处理的sgRNA。将溶解的RNA导入多个管中,然后用光谱仪进行定量。凝胶电泳证实sgRNA的完整性。
3、Cas9m RNA合成:
从Addgene(质粒#42251)获得的pMLM3613载体合成Cas9mRNA17通过用PMEI限制性内切酶对载体进行线性化,用mMESSAGEmMACHINE 进行凝胶纯化和逆转录酶合成® 7Ultra Kit(Thermo Fischer Scientific)(图1b)。 将合成的Cas9 mRNA用DNaseI处理,去除载体DNA,然后用E-PAP酶进行聚A尾化。用氯化锂沉淀聚A尾cas9 mRNA,溶于无核酸酶的水中。
4、截肢和电穿孔:
选择在标准实验室条件下维持9至12个月大的成年斑马鱼,麻醉和尾鳍的远端半部分在无菌条件下截肢条件。
采用一种新的方法将Cas9和sgRNAmRNA电穿孔到截肢的鳍组织。电穿孔前将sgRNA(50ng/µl)和Cas9 mRNA(250ng/µl)的1:5比例混合,在玻璃微滑片中的两个圆线电极(BTX)之间放置25µl。
截肢的斑马鱼尾鳍组织被垂直放置在电极之间的空间,将麻醉的鱼放在海绵中,使鳍的尖端在电穿孔过程中接触RNA溶液。在电穿孔器(ECM2001,BTX哈佛仪器,USA)中,对具有30ms脉冲长度和3个脉冲重复的FIN组织进行了电穿孔,美国)(图1c)。截肢后立即进行电穿孔,头一次剂量和截肢后24小时为第二次剂量。为了一DPA基因型分析仅给予一剂电穿孔。对每组实验进行对照实验,只电穿孔Cas9mRNA和MilliQ水。所有实验均以复制方式进行(n=3)。动物实验和基因操作的程序已按照细胞和分子生物学中心动物伦理委员会(IAEC/CCMB/协议#)批准的协议进行#50&52/2013).
5、基因型和表型分析:
为了进行表型分析,在截肢和电穿孔后5、7和10天收集鳍组织。用image J软件测量再生鳍组织生长。
为了进行基因型分析,在截肢和电穿孔后1、2和7天收集组织。用TRI试剂从单个翅片组织中提取RNA(Sigma,美国)。用反向转录酶核心试剂盒(Eurogentec,比利时)从单个FIN组织中提取的100µgms总RNA样品中合成了cDNA)。
靶基因、ANXA2a和2b基因以及非靶向基因ANXA1a的验证和表达,利用基因特异性引物对采集的单个FIN组织RNA进行定量RTPCR测定ANXA5b、ANXA13 和ODC6,将基因的表达与对照时间点样本进行比较。
6、蛋白质组学和WB分析:
对截肢和电穿孔后1、2和7天采集的翅片组织进行了基于ITRAQ的定量蛋白质组学分析。从各阶段的集合组织中提取蛋白质,定量,1DE凝胶电泳。对凝胶进行不同分子量范围的分馏,进行凝胶内胰蛋白酶消化。目标时间点样本的Cas9对照、ANXA2a和ANXA2b基因分别用iTRAQ标签114、115、116和117标记。在Q-Exactive质谱仪中对ANXA2a和2b分子量分数对应的肽样品进行MS/MSMS分析。分析该蛋白在1,2和7dpa的时间点表达。对ANXA2进行蛋白质样品的Westernblot分析,ANXA1和ODC蛋白的表达利用各自的小鼠特异性抗体来估计蛋白质的表达。
7、数据分析:
使用MS-Excel对所有从各种测量中获得的数据进行分析,并使用PRISM6软件分析数据的统计意义。
三、结果
1、靶和Cas9mRNA的合成
使用ZiFiT程序设计了7个ANXA2a和2个具有BSaI限制位点的22bp长度的ANXA2b特定目标(表1,图1a)。
所有的目标都被设计成在基因的外显子区域。目标被合成为寡核苷酸,并与它们各自的反义寡核苷酸二聚,并在pDR274载体中克隆(图1b)。
从阳性和证实的克隆中合成了靶位点特异性RNA。从每个目标位点获得的mRNA被汇集为各自的目标基因。 同样,从pMLM3613载体(图1b)中合成Cas9mRNA,并与集合的ANXA2a和2b特异性靶m RNA混合,并在截肢后电穿孔给动物。
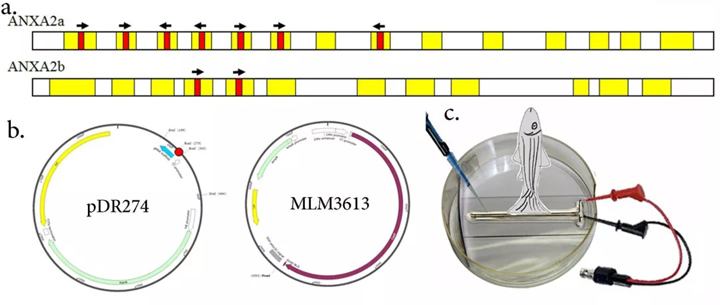
图1:1a.具有外显子(黄色阴影)和CRISPR-Cas目标位点(红色阴影)的ANXA2a和2b基因示意图,箭头指示CRISPRCas目标的方向。 b.DR274质粒图谱,红色点表示克隆位点和MLM3613载体。c.卡通详细介绍了CRISPR-Cas9RNA在成年斑马鱼截肢鳍组织中的电穿孔方案。
2、表型分析
根据斑马鱼尾鳍组织截肢和电穿孔后的表型分析,发现CRISPR靶点对鳍组织的生长有明显的影响(图2)。
从5dpa开始,ANXA2a和2b靶向鳍组织的再生生长缓慢,鳍组织的结构再生明显减少(图2b)。比较翅片长度的生长表现出较强的相关性抑制Annexin基因靶向FIN组织对对照和CAS9靶向再生组织的生长。
此外,再生鳍组织的形状由于ANXA2a和2b目标在截肢后的后期时间点丢失(图2a)。
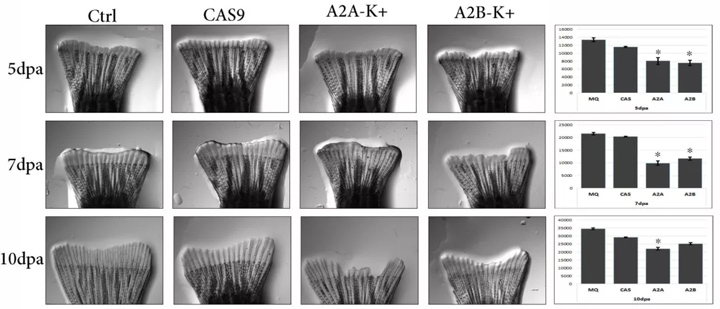
图2:2a.用MQ、Cas9、ANXA2a和ANXA2bCRISPR靶电穿孔5、7和10dpa再生尾鳍组织的表型。 b.条形图详细说明了鳍组织在各自时间点和电穿孔的生长速率。翅片生长为mean+SEM;与MQ-Ctrl组比较*p<0.05。(配对t检验,N=3)
3、基因型分析
在1、2和7dpa时,ANXA2a和2b基因的表达在相应的基因敲除FIN组织中被下调,对CAS9和MQ控制组织有抑制作用(图3a)。
靶区特异性引物扩增的ANXA2a基因的表达显示基因表达完全减少或截短的PCR扩增子,验证了阳性CRISPR基因的作用(图3a,3b)。同样,ANXA2B 基因扩增也被发现在ANXA2B靶向鳍组织中被下调。在这两个基因靶向组织中,发现基因的表达在7dpa处被恢复。
与对照组和正常组织基因表达相比,ANXA2a的表达效果优于ANXA2b基因。有趣的是,ANXA2b的表达在ANXA2a靶向鳍组织中也被发现下调,反之亦然。其他Annexin家族基因的表达分析,如ANXA1a、5b和13,也发现在ANXA2a和2b的敲除组织中,在1和2dpa中被下调。所有Annexin基因在7dpa处均完全恢复基因表达(图3a,3b)。在7dpa时,ANXA2bKD+组织中ANXA2b的表达没有像ANXA2a那样恢复。 在ANXA2a和2b基因敲除后,ANXA2a和2b基因的表达被下调或表达为截短扩增子,证实了CRISPR对靶的作用。
4、蛋白质组学分析
蛋白质组学分析包括Westernblot分析和定量蛋白质组学分析,再生尾鳍组织的ANXA2a和2bCRISPR显示明显下降调节ANXA2和ANXA1蛋白以及其他Annexin家族蛋白。
用ANXA2a和2bCRISPR靶向再生鳍组织中ANXA1和ANXA2蛋白(ANXA2a和2b)表达的Westernblot分析表明,这两种蛋白都有不显著的下调作用(图3c)。
基于TRAQ的与Annexin家族蛋白匹配的蛋白质组分的定量蛋白质组学分析表明,ANXA1a、ANXA2a、ANXA5b和ANXA13蛋白在1、2和7dpa处有明显的下调作用(表2,图3d。由于ANXA2a和2b基因像其他附件素一样被敲除,ANXA11b的表达没有被发现被修饰(图3d)。
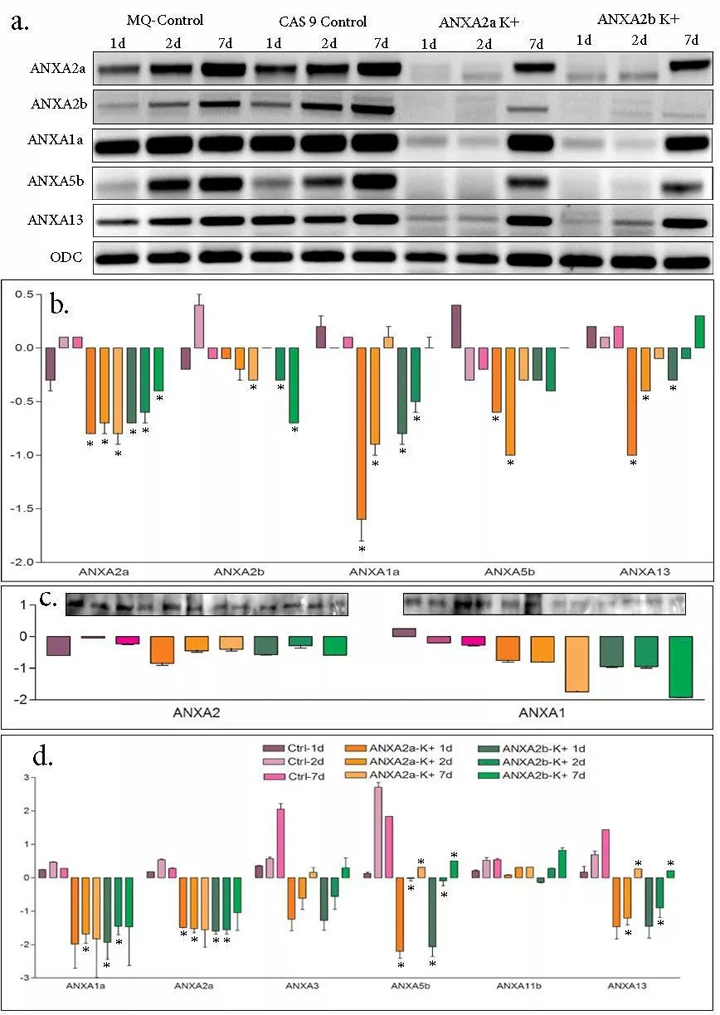
图3:a.基于PCR和凝胶电泳的MQ-Ctrl、CAS9Ctrl、ANXA2aK+和ANXA2bK+FIN组织样品的Annexin基因表达;b.ANXA2a、2b、1a、5b和13个基因的波段强度和条形图。c.ANXA2 和ANXA1蛋白的Westernblot图像及其表达强度。d.基于iTRAQ的Annexin蛋白表达定量蛋白质组学分析。基因和蛋白质的表达表现为mean+SEM;与MQ-Ctrl组比较*p<0.05。(配对t检验,N=3)。
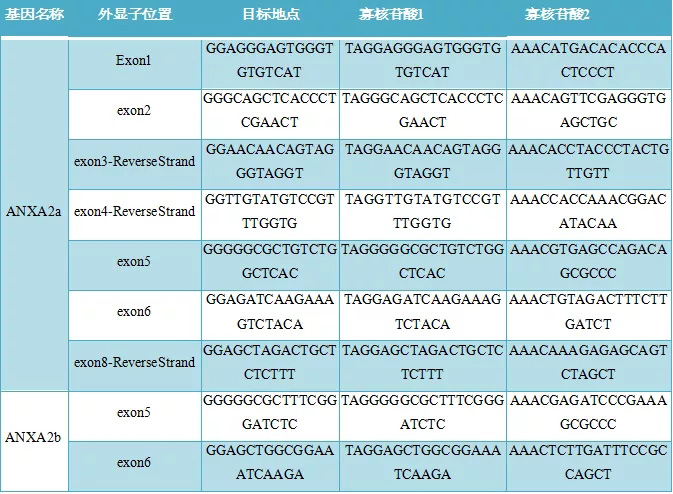
表1:ANXA2a和2b基因特异性CRISPR靶点及其正向和反向寡核苷酸序列列表。

表2:基于iTRAQ的定量蛋白质组学在对照、ANXA2a和ANXA2b靶向斑马鱼尾鳍组织中的表达与各自时间点的MQ-Ctrl样本相比,表达式显示为重复的平均值。
四、讨论
在此,开发并优化了一种基于体内组织特异性电穿孔的新技术,以提高CRISPR在斑马鱼中基因基因组编辑的效率。
Annexin基因有70%的保守性,ANXA2b是人类中唯一不存在的家族成员。我们已经显示了ANXA2a和2b与斑马鱼鳍再生的直接关联。组织特异性基因组编辑效率低下是许多研究者面临的共同问题。
为了解决这一问题,开发和评估了不同的方法。在本研究中,证实了对所有复制没有错配靶向的组织特异性基因组编辑,这表明将Cas9封顶RNA电穿孔到组织中可以减少Cas9翻译的时间,从而提高突变转移到下一代的概率。这种截肢后的电穿孔技术使基因靶向比复杂的基于显微注射的方案容易得多。
因此,这种基于体内组织特异性电穿孔的新技术可以通过CRISPR/Cas9系统提高敲除、敲除和生殖系传输的效率。值得一提的是,在本研究中,ANXA2a和2b的敲除效率得到了提高。这对基因家族其他成员的表达水平有影响,敲除效率是否与Annexin家族的不同基因相关值得进一步研究。选择有效的靶位点和合适的时间点是影响基因编辑成功概率的关键因素。
总之,本研究开发并评价了一种基于体内组织特异性基因电穿孔的新方法。该技术通过使用CRISPR/Cas9系统来提高斑马鱼基因组编辑的效率。这种新的策略将减少不必要的劳动密集型筛选,并将显著提高斑马鱼产生理想突变体的速度。本研究还证实了ANXA2a和ANXA2b是参与再生的重要基因,它与ANXA1、ANXA5和ANXA13基因在斑马鱼尾鳍组织再生中具有相关作用。
参考资料:
[1] Smith, P. & Moss, S. Structural evolution of the annexin supergene family. Trends Genet. 10: 241-246 (1994).
[2] Farber, S. D., De Rose, R. A., Olson, E. S. & Halpren, M. E. The zebrafish annexin gene family. Genome Res. 13: 1082-1096 (2003).
[3] Moss, S. The annexins. In: The annexins (ed. S.E. Moss), pp.1–10. Portland Press, Chapel Hill, NC (1992).
文丨Mir Quoseena,Sowmya Vuppaladadium,Shahid
Hussain, Swarna Bharathi, Mohammed M Idris
翻译丨陈星辉
发表于Biochimie doi:10.1016 / j.biochi.2020.05.014